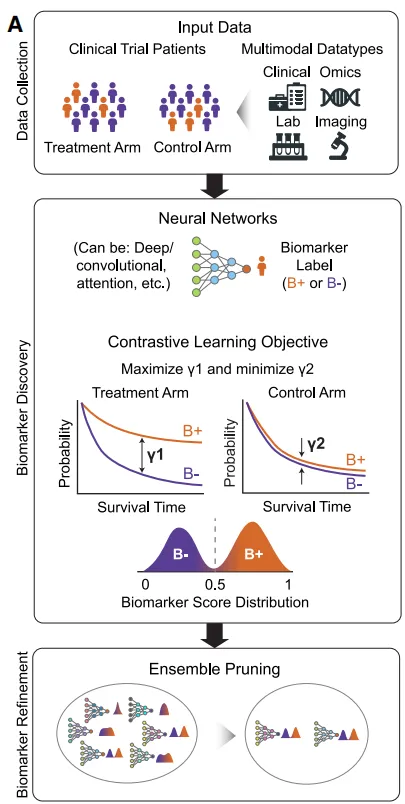
AI-driven predictive biomarker discovery with contrastive learning to improve clinical trial outcomes
作者:
阿斯利康肿瘤研发
Tempus AI 生命科学公司
期刊:cancer cell
重要指数:⭐️⭐️⭐️⭐️⭐️
时间:25.5.12
关键词:虚拟细胞\人工智能
摘要:现代临床试验可对每个个体进行数万次临床基因组学检测。与预后标志物相比,预测性生物标志物的发现仍具挑战性。为解决这一问题,我们提出了一种基于对比学习的神经网络框架——预测性生物标志物建模框架(PBMF),该框架以自动化、系统化且无偏倚的方式探索潜在预测性生物标志物。通过回顾性应用该算法于真实临床基因组学数据集(特别是免疫肿瘤学(IO)试验),我们的算法可识别接受IO治疗且生存期长于其他疗法患者的生物标志物。我们展示了该框架如何通过仅基于早期研究数据揭示可预测且可解释的生物标志物,为一项III期临床试验提供回顾性支持。携带该预测性生物标志物的患者与原始试验相比,生存风险降低15%。 PBMF 为生物标志物策略提供了通用、快速且稳健的方法,为临床决策提供可操作性结果。
1.研究背景
-
为什么一定要预测性,而非预后性:精准医学的核心是给“对”的病人“对”的药;预测性生物标志物(Predictive Biomarker)是衡量“对”与“不对”的标尺。与预后性标志物不同,预测性标志物必须能区分“治疗特异性获益”,否则会把“本身病程温和”误当成“药物有效”,导致 III 期试验失败率居高不下(肿瘤领域≈90%)。传统 Cox-PH、logistic 回归一次只能检验少量变量与交互项,面对高维组学数据时假设空间爆炸,无法无偏地搜索“非线性+多变量组合”的预测信号。
-
“多组学+非线性”已成刚需,但现有机器学习工具仍把“预测性”当后处理。新一代测序使每位病人携带 >10 000 维特征(突变、表达、影像、临床),生物系统冗余导致“多组不同特征可同样预测”。免疫肿瘤药物调节免疫系统而非直接杀瘤,缺乏像 HER2、BCR-ABL 那样单基因假设,必须依赖“多变量复合标志物”。复合生物标志物(即多种临床测量指标的潜在非线性组合)近期被提出用于改善IO的治疗结局预测。现有 subgroup 发现方法(SIDES、Virtual Twins)要么只能做线性/树形拆分,要么把“治疗臂”与“对照臂”分开建模,再事后找切点,无法在同一损失函数里强制“治疗有效 & 对照无效”的对比约束,导致容易拣到预后信号。
-
为解决这些局限性,我们提出预测性生物标志物建模框架(PBMF),这是一种由新型训练目标引导的神经网络驱动对比学习过程。本文提供了大量实证证据,展示 PBMF 在不同场景下(包括模拟生物标志物发现、成熟的生存分析临床数据集以及各类免疫疗法的真实世界和随机对照临床试验数据)的稳健预测性生物标志物发现能力。值得注意的是, PBMF 在模拟和真实数据集中的亚组识别表现均优于现有方法。
-
免疫疗法加速审批路径下,大量 II 期试验为单臂设计,缺乏同期对照,传统方法无法定义“预测性”。FDA 鼓励在特定场景使用外部或合成对照臂(Synthetic Control Arm, SCA),但如何确保 SCA 与试验人群可比、如何从中提取“治疗特异性”信号,尚无标准化统计框架。合成对照臂人群与单臂病人常存在系统差异(基线年龄、既往线数、分子谱),直接把两组拼在一起会引入混杂;需要一种能在“非随机”数据里仍锁定“治疗-交互”信号的学习策略。
2.研究框架
PBMF:通过治疗与对照效应的比值,实现了对治疗特异性预测信号(而非预后信号)的直接学习。
该 PBMF 利用从两个治疗组中每个样本采集的任何模态数据。 PBMF 训练一组神经网络集成模型,每个模型均独立基于临床试验数据使用对比损失函数进行训练。该损失函数旨在增强治疗组中B+与B–的差异性影响,同时最小化对照组中B+对B–的影响。通过剪枝处理, PBMF 模型集成仅保留性能相似的模型。最终输出的整合生物标志物评分仅针对目标治疗的更长事件时间(如生存期)进行富集。
图 1B:模拟数据集上的 Precision-Recall 曲线(AUPRC)比较
结论:PBMF 的 AUPRC = 0.918 ± 0.047,显著高于 Virtual Twins (0.858 ± 0.029) 和 SIDES(基本失败),说明在“已知答案”的合成数据里 PBMF 能更准确地找回真正的预测性信号。
图C:PBMF 、VT和SIDES方法在全部9个测试数据集及各生物标志物状态(B+和B–)下治疗方案的风险比。
阴影区域对应各方法在特定生物标志物状态(B+和B–)下最大与最小风险比定义的边界框。PBMF 在持续识别预测性生物标志物方面显著优于其他方法
图D:森林图展示 PBMF 、VT和SIDES方法在测试数据集上的性能比较(公开经典临床数据集(乳腺癌 + 糖尿病视网膜))
森林图左侧标注患者数量(N),其中TRT=目标预测性生物标志物的治疗方案(如颞叶的IO), CTL =对照治疗方案(如颞叶的化疗)。把图 1C 的观察量化,PBMF 在所有 9 个外部验证中均保持 B+ 显著且 B– 不显著,而竞品至少失败 3-4 次。
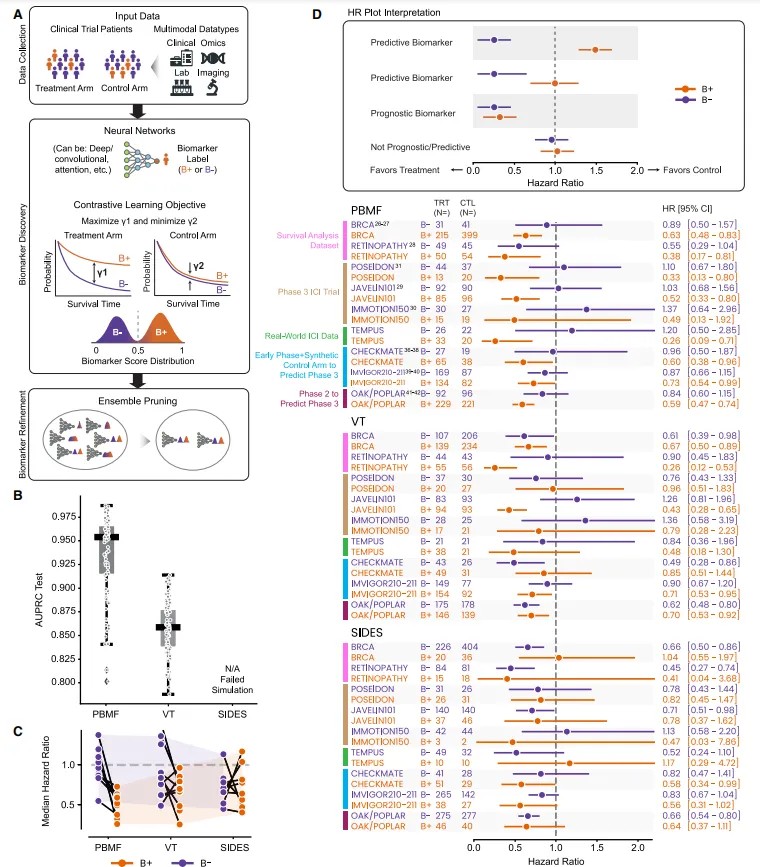
图2A:来自成熟临床数据集的生存分析测试数据(公开经典临床数据集(乳腺癌 + 糖尿病视网膜),同图1D)
-
乳腺癌:PBMF B+ 中位 OS 显著长于 B–(HR 0.63),且两条对照曲线几乎重合;VT 的 B+ 在对照化疗里也一样长,已滑向预后性。
-
糖尿病:PBMF B+ 激光治疗组 5 年视力丧失率 20 %,B– 45 %;对照无治疗组两条曲线重叠,符合预测性定义。
图B:免疫肿瘤学临床试验测试数据(Poseidon, Javelin 101,和IMmotion 150)(图1D中段,图 2B(KM 三套))
-
POSEIDON:仅 PBMF 的 B+ 在 durva+chemo 臂显著延长 OS(HR 0.33),B– 无差异;VT 虽 HR 好看,但 KM 图显示其 B+ 在对照臂也差,实为预后。
-
JAVELIN 101:同样只有 PBMF 真正做到“治疗臂分开、对照臂重合”。(VT研究似乎也发现了一个普适性预测性生物标志物(B+组:HR=0.43,CI=0.28–0.65,p=5.48e-5;B–组:HR=1.26,CI=0.81–1.96,p=3.10e-1),但Kaplan-Meier曲线分析表明,其识别的B+组别接受对照治疗(舒尼替尼)后生存期反而更差)
-
IMmotion150:PBMF 趋势最佳,VT 的 B+ 在对照臂反而更差,再次验证 PBMF 锁定的是“治疗-特异”信号而非一般好预后。(B+组别在所有治疗方案中的生存期趋势均比B–组别更差)
图2C:真实世界数据(Tempus NSCLC)
训练集 VT 也能拉开,但到了外部队列仅 PBMF 的 B+ 在免疫治疗组显著优于化疗(HR 0.26),VT 的 B+ 在两组已无差异,说明 RWD 场景下 PBMF 同样稳健。
图2D:利用合成对照臂的 CheckMate 系列与 IMvigor 系列 KM 曲线。在 CheckMate 和 IMvigor 211 试验中,PBMF 和 VT 都成功地把“预测性生物标志物”从训练集外推到验证集,而 SIDES 没有。
IMvigor 211 是一个 III 期试验,比较 atezolizumab(免疫药) vs 化疗 用于晚期尿路上皮癌。
-
训练集 用了:
-
IMvigor 210(单臂 II 期,只有 atezo)
-
从 IMvigor 211 中随机抽的 100 例化疗患者(这叫“合成对照臂”)
-
-
验证集 是 IMvigor 211 中 剩下的 472 例患者(排除那 100 例化疗患者)。
-
IMvigor 211 官方 BEP 人群的 HR = 0.81(atezo vs 化疗)。PBMF 把 HR 从 0.81 → 0.73,风险降低 ≈ (0.81-0.73)/0.81 ≈ 10%。VT 从 0.81 → 0.71,风险降低 ≈ 12%。
结论:
-
CheckMate-010/025:PBMF 在验证集 HR=0.60,比原始 ITT HR=0.73 再降 12 %;VT 则出现 B+ 在对照臂更优的“反向”结果。
-
IMvigor210/211:PBMF 与 VT 均达到预测性,但 PBMF 风险降幅更大(10 % vs 12 %),SIDES 再次 prognostic。
图2E:OAK42三期临床试验测试数据集。时间轴以月为单位。
如果不用“单臂+合成对照”,而用 双臂 II 期(POPLAR) 训练,再去预测 III 期 OAK:PBMF 在 OAK 的 B+ HR = 0.59(比 OAK 官方 0.65 又降了 9%)。而VT 和 SIDES 这次只找到“预后性”标志物(B+ 和 B– 在两种治疗里都获益或都吃亏,无法区分谁该用 atezo)。
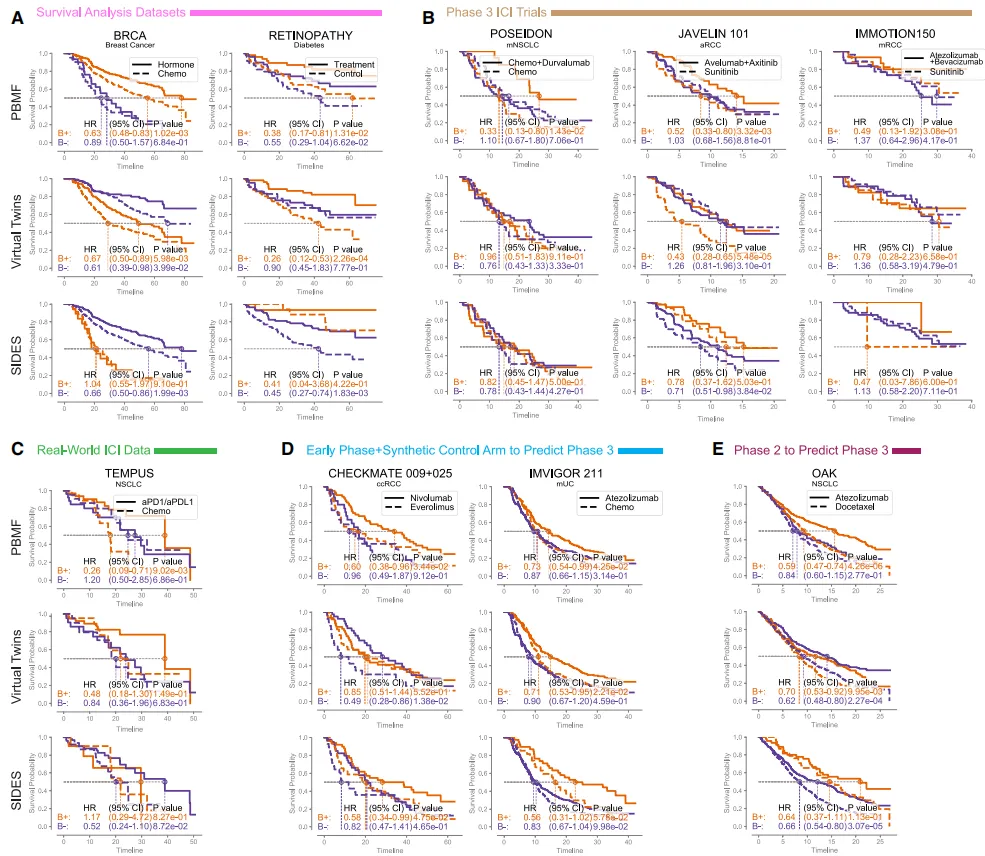
图3A:从黑箱到白箱——模型蒸馏流程
通过从修剪后的 PBMF 集成生成的生物标志物评分进行伪标签化,可识别出高置信度患者样本。这些样本随后用于构建可解释的简化决策树模型,将患者分类为B+或B–
-
33 个小神经网络(PBMF ensemble)→ 输出 0–1 的“生物标志物分数”。
-
用 0.5 ± ε 的阈值把“高置信”样本挑出来(红色 = B+,蓝色 = B–),这些标签叫“伪标签”。
-
用伪标签去训练一棵决策树(depth ≤ 5),得到 if-else 规则。
结论:把“深度学习”变成“医生能看懂的 5 层问题”。
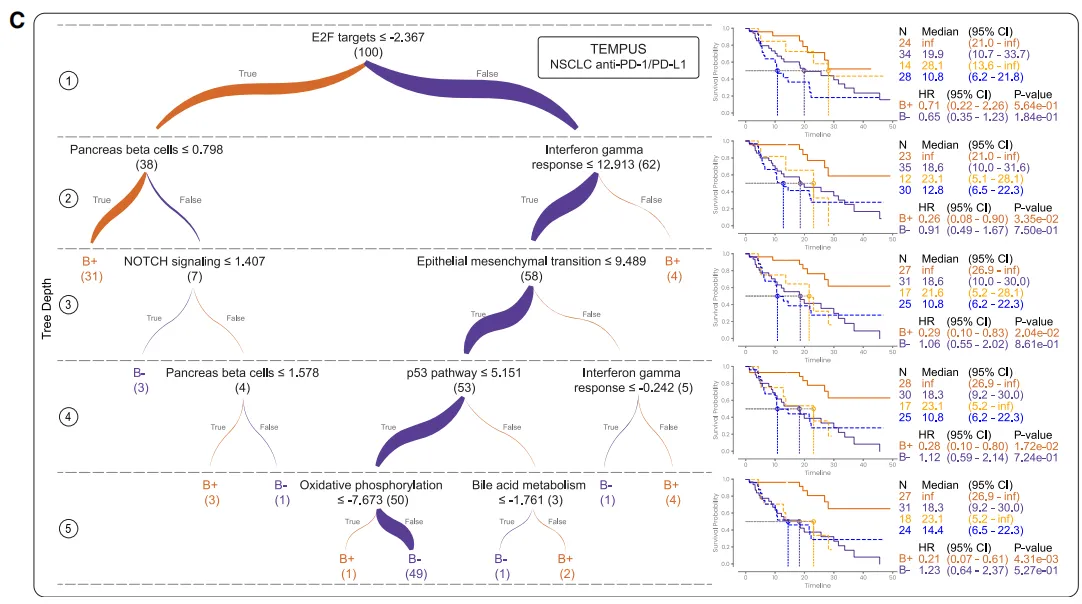
图3B:从POSEIDON31训练数据生成的蒸馏 PBMF 决策树示例(深度=5)
左(树结构):叶子节点的(10)、(104)指人数
右(KM 曲线):在每层的B+ vs B–
结论:
-
树越深,HR 越小(0.48 → 0.21),但阳性人数几乎不变 → 信号更纯,可读性仍在。
-
生物学解释:外周血里“巨噬细胞少 + B 细胞多”提示肿瘤微环境更“热”,免疫治疗获益大。
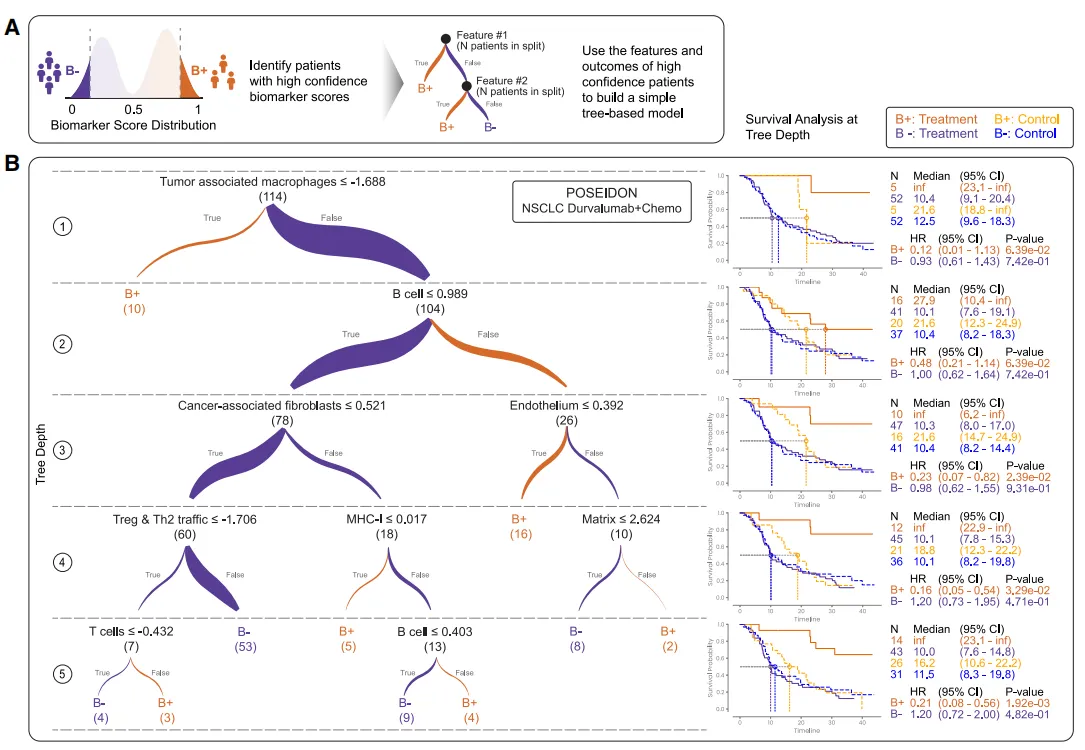
图3C:从tempus真实世界训练数据生成的蒸馏 PBMF 决策树示例(深度=5)
-
先用“低增殖 + 高 IFN-γ”把 prognostic 信号洗掉,再细化得到纯预测性规则。
-
虽然生物学解释不如 3B 直白,但同样能用 5 层树把疗效差异放大。
我们设计了一种端到端的生物标志物发现流程,该流程可生成易于人类理解的预测性生物标志物原型,为临床应用做好准备。
A:临床试验数据与终点收集:发现集(Poplar 2期临床试验41)数据集的Kaplan-Meier曲线。图中提供了各试验中生物标志物阳性/阴性组及治疗/对照组的样本量(N)。
4B – 预测性生物标志物的识别:利用发现集(Poplar试验), PBMF 成功发现了一种可识别哪些患者在阿特珠单抗治疗下生存期更长(而非多西他赛)的生物标志物。
4C – 预测性生物标志物的优化:该优化涉及从集成模型中剪枝以消除虚假模型
4D – 随后推导出一个规则集(即决策树),以封装该生物标志物的预测能力。线条粗细与括号内患者数量成正比。
4E – 独立验证集:OAK 3期试验42(上图)及使用Poplar试验中识别的简化预测性生物标志物对OAK试验进行患者分层(下图)。这种 PBMF 模型的独立数据集验证证实了生物标志物的预测能力,表明该模型从集成到简化树状表示均具有可靠性,从而强化了其在临床试验分层中的应用价值。
4F – 与单变量标志物对比(图 4F + 表 S5)
-
血 TMB(bTMB)单变量: – 训练集 HR = 0.78,验证集 HR = 0.71
-
PBMF 复合树: – 训练集 HR = 0.46,验证集 HR = 0.55 👉 复合标志物把风险降得更低,且阳性率更高。
一眼读:
-
4A→4B:训练集已见 B+ 红实 显著优于 B– 蓝实。
-
4C:剪枝后 HR 中心值略降、误差线缩短→方差减小。
-
4D:7 个变量、5 层、64 % 阳性率,医生可读。
-
4E:同规则在 OAK HR=0.55,比 ITT 再降 15 %。
-
4F:复合标志物在两套数据均显著优于传统血 TMB,证明“多变量非线性”必要。
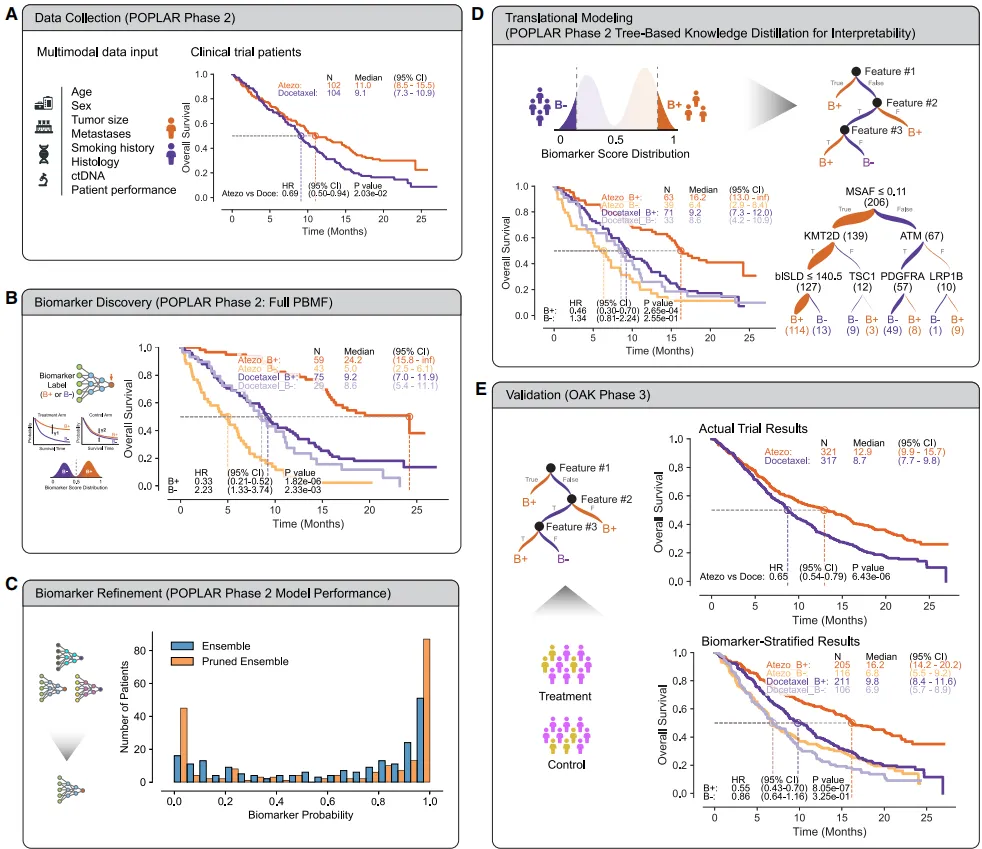
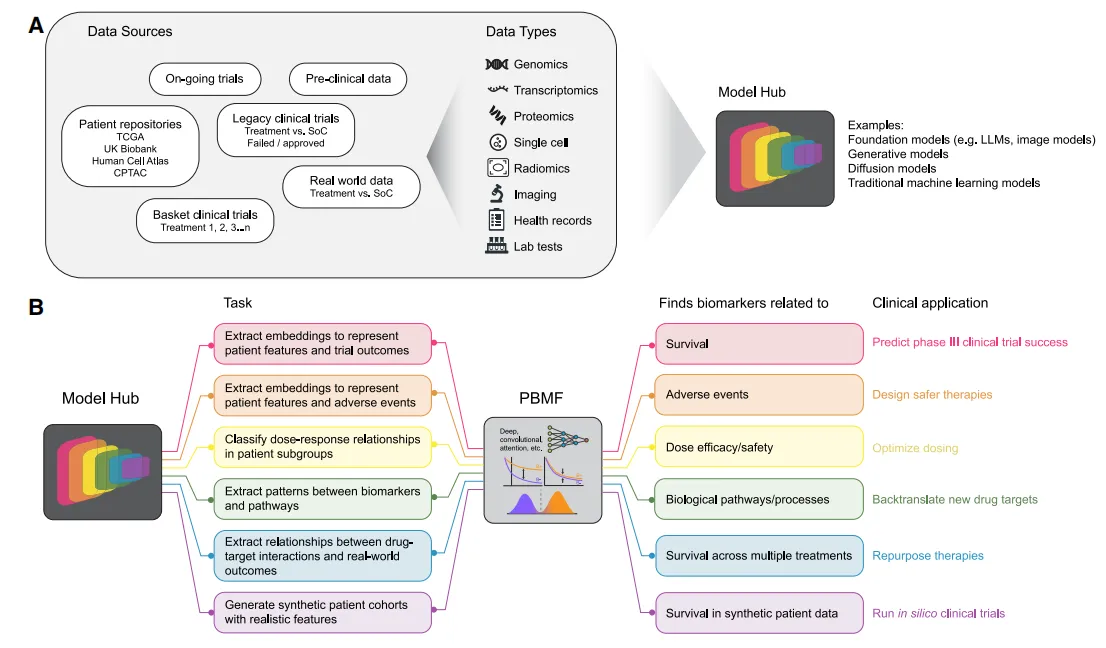
图5:PBMF 的潜在未来应用场景
(A)该 PBMF 是一个端到端的应用程序编程接口(API),能够整合跨多种数据模态的预训练模型,例如基因组数据、放射组学数据和临床数据预测性生物标志物发现的数据来源包括患者数据库(如 TCGA 和英国生物样本库)、各类临床试验(如既往失败试验、多治疗篮试验)以及真实世界数据。这些数据源涵盖基因组学、放射组学、影像学和健康记录等多种数据类型,可输入到包含多种预训练模型的“模型中心”,包括大语言模型、生成模型、扩散模型和传统机器学习模型。
(B)PBMF 的模块化特性得益于其与多种可微分模型的兼容性,能够处理这些嵌入数据以发现生存期、不良事件、给药策略等生物标志物。该 PBMF 利用预训练模型生成的嵌入式数据、模式和/或合成数据,这些模型经过针对特定任务的微调。
讨论与不足
PBMF 为什么赢:
横跨 9 个不同来源、不同癌种、不同模态的数据集,PBMF 在模拟到 III 期试验中均优于现有方法,唯一稳定输出预测性信号。其对比损失同时放大治疗差异、压平对照差异,避免预后混淆;蒸馏后 5 层决策树可直接用于患者筛选,传统 Cox 模型要穷举所有变量组合,而 PBMF 用梯度下降自动搜索,既省人力又避免漏掉高阶交互。
-
方法学拆解:三条设计决策的价值 内容 a) 比例型损失 → 强制网络同时优化“拉大治疗、压小对照”,避免事后切点。 b) 集成 Bagging+剪枝 → 在小样本高维场景降低方差,且无需交叉验证调参。 c) 蒸馏决策树 → 把 ensemble 黑箱转成 5 层深度内的白箱,保留 90 % 以上预测力,可直接写进试验方案或监管递交材料。
-
临床转化示例:如何用早期数据影响 III 期 内容 • 以 POPLAR→OAK 为例,展示“II 期数据→蒸馏规则→III 期验证”完整闭环:死亡风险再降 15 %,阳性率仍保持 64 %,具备可操作的商业规模。 • 指出这是首个“仅用单臂/小样本早期数据+历史对照”就能 retrospectively 提升 III 期疗效的研究,为“提前终止/加速审批”提供定量依据。
-
与未来技术栈的嫁接:API 化与多模态 内容 • PBMF 被封装成可插拔 API,可接受任何可微模型(CNN、Transformer、GNN)输出的 embedding,因此能无缝吸收影像、病理、文本等预训练大模型。 • 举例:把病理 ViT 或基因组 LLM 的 embedding 当输入,可扩展至不良反应、剂量-暴露、联合用药等预测任务,成为“精准研发操作系统”的核心模块。
-
局限性与最佳实践:把“不能做什么”说清 内容 • 无法保证在特定队列的可用特征中存在预测信号:信号不存在就别硬找:若特征库只有年龄/性别,任何方法都只能给出预后性。 • 集成 PBMF 可能无法在简化为单一模型时保持其预测效能,因为生物标志物的预测能力与其简洁性之间往往存在权衡。强预后变量可掩盖预测性:建议先进行“预后-正交”预处理或加权。
-
虽然 PBMF 在从噪声或预后特征中识别预测信号方面优于其他方法,但我们仍需注意:强预后特征可能阻碍预测信号的识别,因此我们的方法可能需要通过前期特征筛选获得更大优势。
-
PBMF 的对比损失函数设计容易弱化那些在对照治疗中仅显示温和正向效应、但在目标治疗中具有显著获益的生物标志物发现。
-
PBMF 作为发现工具,任何生物标志物假设都需要前瞻性临床验证。
-
-
结论与下一步 内容 • PBMF 提供“端到端、可解释、外部验证”的预测性标志物发现流水线,在免疫肿瘤和合成对照场景下把疗效数字再抬 10–15 %,具备立即写入临床开发策略的成熟度。 • 未来工作将探索①纯 RWD 合成对照、②多药多臂组合、③前瞻性篮子试验,以进一步检验通用性与监管接受度。